开发科学软件
第二部分:使用Python的实际方面
在本文中,我们将遵循本系列文章的第一部分中介绍的开发科学软件的TDD原则,来开发一种称为Sobel滤波器的边缘检测滤波器。
在第一篇文章中,我们讨论了为科学软件开发可靠的测试方法的重要性和复杂性。我们还看到了如何通过遵循TDD启发的开发周期来克服这些问题,但为科学计算进行了调整。下面是这些指令的缩写版本。
实施周期
- 收集需求
- 草拟设计
- 实施初步测试
- 实施您的Alpha版本
- 构建Oracle库
- 重新审查测试
- 实施性能分析
优化周期
- 优化
- 重新实施
新方法周期
- 实施新方法
- 与先前精心策划的Oracle进行验证
入门:Sobel滤波器
在本文中,我们将按照上述指示开发一个应用Sobel滤波器的函数。Sobel滤波器是一种常用的计算机视觉工具,用于检测或增强图像中的边缘。继续阅读以查看一些示例!
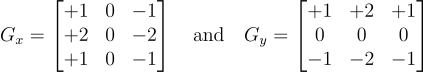
从第一步开始,我们收集一些需求。我们将遵循本文中描述的Sobel滤波器的标准公式。简单来说,Sobel算子由将图像A与以下两个3×3卷积核进行卷积,对结果图像中的每个像素进行平方,求和并逐点进行平方根运算组成。如果Ax和Ay是卷积的结果图像,则Sobel滤波器S的结果为√(Ax² + Ay²)。
要求
我们希望这个函数接受任何2D数组并生成另一个2D数组。我们可能希望它在ndarray的任何两个轴上操作。甚至我们可能希望它在超过两个轴上(或少于两个轴上)工作。我们可能对如何处理数组的边缘有规格要求。
现在,让我们记住保持简单,并从2D实现开始。但在此之前,让我们草拟设计。
草拟设计
我们从一个简单的设计开始,利用Python的注解。我强烈推荐尽可能多地使用注解,并使用mypy作为linter。
from typing import Tuplefrom numpy.core.multiarray import normalize_axis_indexfrom numpy.typing import NDArraydef sobel(arr: NDArray, axes: Tuple[int, int] = (-2, -1)) -> NDArray: """计算图像的Sobel滤波器 参数 ---------- arr : NDArray 输入图像 axes : Tuple[int, int], 可选 要计算滤波器的轴,默认为(-2,-1) 返回 ------- NDArray 输出 """ # 仅接受2D数组 if arr.ndim != 2: raise NotImplementedError # 确保axis[0]是第一个轴,axis[1]是第二个轴 # 这个不常见的`normalize_axis_index`将负索引转换为0到arr.ndim - 1之间的索引。 if any( normalize_axis_index(ax, arr.ndim) != i for i, ax in zip(range(2), axes) ): raise NotImplementedError pass实现测试
这个函数并没有做太多的事情。但是它有文档、注释,并且当前的限制已经嵌入其中。现在我们有了一个设计,我们立即转向测试。
首先,我们注意到空图像(全零)没有边缘。因此它们也必须输出零。事实上,任何常数图像也应该返回零。让我们将这些内容融入到我们的第一个测试中。我们还将看到如何使用猴子测试来测试多个数组。
# test_zero_constant.pyimport numpy as npimport pytest# 同时测试多个数据类型@pytest.mark.parametrize( "dtype", ["float16", "float32", "float64", "float128"],)def test_zero(dtype): # 设置随机种子 seed = int(np.random.rand() * (2**32 - 1)) np.random.seed(seed) # 创建一个随机形状的二维数组,并用零填充 nx, ny = np.random.randint(3, 100, size=(2,)) arr = np.zeros((nx, ny), dtype=dtype) # 应用sobel函数 arr_sob = sobel(arr) # `assert_array_equal` 大多数情况下会失败。 # 它只有在 `arr_sob` 完全等于零时才能通过, # 这通常不是情况。 # 不要使用! # np.testing.assert_array_equal( # arr_sob, 0.0, err_msg=f"{seed=} {nx=}, {ny=}" # ) # `assert_almost_equal` 在使用高小数位数时可能失败。 # 它还依赖于float64的检查,对于float128类型可能会失败。 # 不要使用! # np.testing.assert_almost_equal( # arr_sob, # np.zeros_like(arr), # err_msg=f"{seed=} {nx=}, {ny=}", # decimal=4, # ) # 使用自定义容差的 `assert_allclose` 是我首选的方法 # 这里的10是任意选择的,取决于具体问题。如果你知道一个方法是正确的,但测试未通过,请增加到100等。 # 如果为了使测试通过所需的容差太大,则确保该方法实际上是正确的。 tol = 10 * np.finfo(arr.dtype).eps err_msg = f"{seed=} {nx=}, {ny=} {tol=}" # 记录种子和其他信息 np.testing.assert_allclose( arr_sob, np.zeros_like(arr), err_msg=err_msg, atol=tol, # rtol 对于期望值等于零是无用的 )@pytest.mark.parametrize( "dtype", ["float16", "float32", "float64", "float128"])def test_constant(dtype): seed = int(np.random.rand() * (2**32 - 1)) np.random.seed(seed) nx, ny = np.random.randint(3, 100, size=(2,)) constant = np.random.randn(1).item() arr = np.full((nx, ny), fill_value=constant, dtype=dtype) arr_sob = sobel(arr) tol = 10 * np.finfo(arr.dtype).eps err_msg = f"{seed=} {nx=}, {ny=} {tol=}" np.testing.assert_allclose( arr_sob, np.zeros_like(arr), err_msg=err_msg, atol=tol, # rtol 对于期望值等于零是无用的 )可以通过命令行运行以下代码片段:
$ pytest -qq -s -x -vv --durations=0 test_zero_constant.pyAlpha 版本
当然,我们的测试当前会失败,但现在已经足够了。让我们实现第一个版本。
from typing import Tupleimport numpy as npfrom numpy.core.multiarray import normalize_axis_indexfrom numpy.typing import NDArraydef sobel(arr: NDArray, axes: Tuple[int, int] = (-2, -1)) -> NDArray: if arr.ndim != 2: raise NotImplementedError if any( normalize_axis_index(ax, arr.ndim) != i for i, ax in zip(range(2), axes) ): raise NotImplementedError # 定义滤波器核。注意它们继承了输入类型,所以 # float32 输入永远不需要转换为 float64 进行计算。 # 但是你能看到在某些输入数据类型下,对 Gx 和 Gy 使用 # 另一个 dtype 可能是有意义的吗? Gx = np.array( [[-1, 0, 1], [-2, 0, 2], [-1, 0, 1]], dtype=arr.dtype, ) Gy = np.array( [[-1, -2, -1], [0, 0, 0], [1, 2, 1]], dtype=arr.dtype, ) # 创建输出数组并用零填充 s = np.zeros_like(arr) for ix in range(1, arr.shape[0] - 1): for iy in range(1, arr.shape[1] - 1): # 点乘后求和,也就是卷积 s1 = (Gx * arr[ix - 1 : ix + 2, iy - 1 : iy + 2]).sum() s2 = (Gy * arr[ix - 1 : ix + 2, iy - 1 : iy + 2]).sum() s[ix, iy] = np.hypot(s1, s2) # np.sqrt(s1**2 + s2**2) return s有了这个新功能,我们所有的测试都应该通过,我们应该得到以下输出:
$ pytest -qq -s -x -vv --durations=0 test_zero_constant.py........======================================== 最慢的持续时间 =========================================0.09s 调用 t_049988eae7f94139a7067f142bf2852f.py::test_constant[float16]0.08s 调用 t_049988eae7f94139a7067f142bf2852f.py::test_zero[float64]0.06s 调用 t_049988eae7f94139a7067f142bf2852f.py::test_constant[float128]0.04s 调用 t_049988eae7f94139a7067f142bf2852f.py::test_zero[float128]0.04s 调用 t_049988eae7f94139a7067f142bf2852f.py::test_constant[float64]0.02s 调用 t_049988eae7f94139a7067f142bf2852f.py::test_constant[float32]0.01s 调用 t_049988eae7f94139a7067f142bf2852f.py::test_zero[float16](17 个持续时间 < 0.005s 隐藏。使用 -vv 显示这些持续时间。)8 个通过,在 0.35s 内到目前为止,我们已经:
- 收集了我们的问题要求。
- 勾勒了一个初步的设计。
- 实现了一些测试。
- 实现了 alpha 版本,通过了这些测试。
这些测试为未来版本提供了验证,以及一个非常基本的 oracle 库。但是,虽然单元测试非常擅长捕捉与预期结果的微小偏差,但它们并不擅长区分错误的结果和定量上不同但仍然正确的结果。假设明天我们想要实现另一个具有更长卷积核的 Sobel 类型算子。我们不希望结果与当前版本完全匹配,但我们希望两个函数的输出在质量上非常相似。
此外,尝试使用许多不同的输入来测试我们的函数是非常好的实践,以了解它们对不同输入的行为。这样可以确保我们对结果进行科学验证。
Scikit-image 拥有一套出色的图像库,我们可以用它来创建 oracle。
# !pip installscikit-image poochfrom typing import Dict, Callableimport numpy as npimport skimage.databwimages: Dict[str, np.ndarray] = {}for attrname in skimage.data.__all__: attr = getattr(skimage.data, attrname) # Data are obtained via function calls if isinstance(attr, Callable): try: # Download the data data = attr() # Ensure it is a 2D array if isinstance(data, np.ndarray) and data.ndim == 2: # Convert from various int types to float32 to better # assess precision bwimages[attrname] = data.astype(np.float32) except: continue# Apply sobel to imagesbwimages_sobel = {k: sobel(v) for k, v in bwimages.items()}一旦我们有了数据,我们可以绘制它。
import matplotlib.pyplot as pltfrom mpl_toolkits.axes_grid1 import make_axes_locatabledef create_colorbar(im, ax): divider = make_axes_locatable(ax) cax = divider.append_axes("right", size="5%", pad=0.1) cb = ax.get_figure().colorbar(im, cax=cax, orientation="vertical") return cax, cbfor name, data in bwimages.items(): fig, axs = plt.subplots( 1, 2, figsize=(10, 4), sharex=True, sharey=True ) im = axs[0].imshow(data, aspect="equal", cmap="gray") create_colorbar(im, axs[0]) axs[0].set(title=name) im = axs[1].imshow(bwimages_sobel[name], aspect="equal", cmap="gray") create_colorbar(im, axs[1]) axs[1].set(title=f"{name} sobel")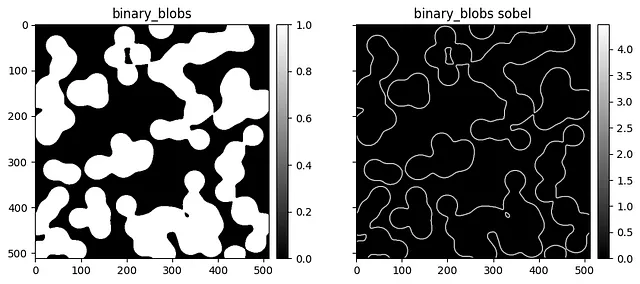

看起来非常好!我建议将它们存储起来,既可以作为数组,也可以作为图像,这样我可以快速对比新版本。我强烈推荐使用HD5F进行数组存储。您还可以设置一个Sphinx Gallery,以便在更新文档时直接生成这些图像,这样您就拥有可重现的图像构建,可以与之前的版本进行对比。
在结果经过验证后,您可以将它们存储在磁盘上,并将其用作单元测试的一部分。类似下面这样:
oracle_library = [(k, v, bwimages_sobel[k]) for k, v in bwimages.items()]# save_to_disk(oracle_library, ...)
# test_oracle.pyimport numpy as npimport pytestfrom numpy.typing import NDArray# oracle_library = read_from_disk(...)@pytest.mark.parametrize("name,input,output", oracle_library)def test_oracles(name: str, input: NDArray, output: NDArray): output_new = sobel(input) tol = 10 * np.finfo(input.dtype).eps mean_avg_error = np.abs(output_new - output).mean() np.testing.assert_allclose( output_new, output, err_msg=f"{name=} {tol=} {mean_avg_error=}", atol=tol, rtol=tol, )性能分析
对这些数据集进行Sobel滤波花费了一些时间!因此,下一步是对代码进行性能分析。我建议为每个测试创建一个benchmark_xyz.py文件,并定期重新运行它们以检测性能回归。这甚至可以作为您的单元测试的一部分,但在本例中我们不会做得那么深入。另一个想法是使用上面的oracle进行基准测试。
有很多计时代码执行的方法。使用内置的time模块中的perf_counter_ns来测量纳秒级的系统整体实际耗时。在Jupyter notebook中,内置的%%timeit魔法命令可以计时特定单元格的执行时间。下面的装饰器受到这个cell magic的启发,可以用于计时任何函数。
import timefrom functools import wrapsfrom typing import Callable, Optionaldef sizeof_fmt(num, suffix="s"): for unit in ["n", "μ", "m"]: if abs(num) < 1000: return f"{num:3.1f} {unit}{suffix}" num /= 1000 return f"{num:.1f}{suffix}"def timeit( func_or_number: Optional[Callable] = None, number: int = 10,) -> Callable: """将其应用于函数以计时其执行时间。 参数 ---------- func_or_number : Optional[Callable], optional 要装饰的函数或`number`参数(参见下文),默认为None number : int, optional 函数运行的次数以获取统计数据, 默认为10 返回 ------- Callable 当提供一个函数时,返回装饰后的函数。否则返回装饰器。 示例 -------- .. code-block:: python @timeit def my_fun(): pass @timeit(100) def my_fun(): pass @timeit(number=3) def my_fun(): pass """ if isinstance(func_or_number, Callable): func = func_or_number number = number elif isinstance(func_or_number, int): func = None number = func_or_number else: func = None number = number def decorator(f): @wraps(f) def wrapper(*args, **kwargs): runs_ns = np.empty((number,)) # 首次运行并测量结果 start_time = time.perf_counter_ns() result = f(*args, **kwargs) runs_ns[0] = time.perf_counter_ns() - start_time for i in range(1, number): start_time = time.perf_counter_ns() f(*args, **kwargs) # 没有存储,更快 runs_ns[i] = time.perf_counter_ns() - start_time time_msg = f"{sizeof_fmt(runs_ns.mean())} ± " time_msg += f"{sizeof_fmt(runs_ns.std())}" print( f"{time_msg} 每次运行(平均 ± {number} 次运行的标准差)" ) return result return wrapper if func is not None: return decorator(func) return decorator将我们的函数进行测试:
arr_test = np.random.randn(500, 500)sobel_timed = timeit(sobel)sobel_timed(arr_test);# 3.9s ± 848.9 ms per run (mean ± std. dev. of 10 runs)速度不太快 🙁
在调查慢速或性能下降时,还可以跟踪每行的运行时间。 line_profiler 库非常适合这个任务。可以通过 Jupyter 单元格魔法或使用该 API 来使用它。下面是一个 API 示例:
from line_profiler import LineProfilerlp = LineProfiler()lp_wrapper = lp(sobel)lp_wrapper(arr_test)lp.print_stats(output_unit=1) # 1 代表秒,1e-3 代表毫秒,等等。下面是一个示例输出:
Timer unit: 1 sTotal time: 4.27197 sFile: /tmp/ipykernel_521529/1313985340.pyFunction: sobel at line 8Line # Hits Time Per Hit % Time Line Contents============================================================== 8 def sobel(arr: NDArray, axes: Tuple[int, int] = (-2, -1)) -> NDArray: 9 # 仅接受 2D 数组 10 1 0.0 0.0 0.0 if arr.ndim != 2: 11 raise NotImplementedError 12 13 # 确保 axis[0] 是第一个轴,axis[1] 是第二个轴 14 # axis。obscure 的 `normalize_axis_index` 将负索引转换为 15 # 0 到 arr.ndim - 1 之间的索引。 16 1 0.0 0.0 0.0 if any( 17 normalize_axis_index(ax, arr.ndim) != i 18 1 0.0 0.0 0.0 for i, ax in zip(range(2), axes) 19 ): 20 raise NotImplementedError 21 22 1 0.0 0.0 0.0 Gx = np.array( 23 1 0.0 0.0 0.0 [[-1, 0, 1], [-2, 0, 2], [-1, 0, 1]], 24 1 0.0 0.0 0.0 dtype=arr.dtype, 25 ) 26 1 0.0 0.0 0.0 Gy = np.array( 27 1 0.0 0.0 0.0 [[-1, -2, -1], [0, 0, 0], [1, 2, 1]], 28 1 0.0 0.0 0.0 dtype=arr.dtype, 29 ) 30 1 0.0 0.0 0.0 s = np.zeros_like(arr) 31 498 0.0 0.0 0.0 for ix in range(1, arr.shape[0] - 1): 32 248004 0.1 0.0 2.2 for iy in range(1, arr.shape[1] - 1): 33 248004 1.8 0.0 41.5 s1 = (Gx * arr[ix - 1 : ix + 2, iy - 1 : iy + 2]).sum() 34 248004 1.7 0.0 39.5 s2 = (Gy * arr[ix - 1 : ix + 2, iy - 1 : iy + 2]).sum() 35 248004 0.7 0.0 16.8 s[ix, iy] = np.hypot(s1, s2) 36 1 0.0 0.0 0.0 return s大部分时间都花在最内层的循环上。NumPy 更喜欢向量化数学运算,因为它可以依赖编译后的代码。由于我们使用了显式的 for 循环,所以这个函数非常慢。
此外,在循环内部进行内存分配是很重要的。NumPy在循环内部分配小量内存时是比较聪明的,所以定义s1或s2的代码行可能并不会分配新的内存。但它们也可能会分配内存,或者可能会发生一些我们不知道的其他内存分配操作。因此,我建议也进行内存分析。我喜欢使用Memray进行分析,但还有其他工具如Fil和Sciagraph。我也建议避免使用memory_profiler,因为它(非常不幸地!)已经不再维护。而且Memray更加强大。下面是Memray的一个示例:
$ # 创建保存分析信息的sobel.bin$ memray run -fo sobel.bin --trace-python-allocators sobel_run.py将分析结果写入sobel.binMemray警告:将aligned_alloc的符号从0x7fc5c984d8f0更正为0x7fc5ca4a5ce0[memray] 成功生成了分析结果。您现在可以从存储的分配记录中生成报告。生成报告的一些示例命令:python3 -m memray flamegraph sobel.bin
$ # 生成火焰图$ memray flamegraph -fo sobel_flamegraph.html --temporary-allocations sobel.bin⠙ 计算高水位线... ━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━╸ 99% 0:00:0100:01⠏ 处理分配记录... ━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━╸ 98% 0:00:0100:01写入sobel_flamegraph.html
$ # 显示内存树$ memray tree --temporary-allocations sobel.bin⠧ 计算高水位线... ━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━╸ 100% 0:00:0100:01⠧ 处理分配记录... ━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━╸ 100% 0:00:0100:01分配元数据-------------------命令行参数:'memray run -fo sobel.bin --trace-python-allocators sobel_run.py'峰值内存大小:11.719MB分配次数:15332714最大的10个分配:-----------------------📂 123.755MB (100.00 %) <ROOT> └── [[3 frames hidden in 2 file(s)]] └── 📂 123.755MB (100.00 %) _run_code /usr/lib/python3.10/runpy.py:86 ├── 📂 122.988MB (99.38 %) <module> sobel_run.py:40 │ ├── 📄 51.087MB (41.28 %) sobel sobel_run.py:35 │ ├── [[1 frames hidden in 1 file(s)]] │ │ └── 📄 18.922MB (15.29 %) _sum │ │ lib/python3.10/site-packages/numpy/core/_methods.py:49 │ └── [[1 frames hidden in 1 file(s)]] │ └── 📄 18.921MB (15.29 %) _sum │ lib/python3.10/site-packages/numpy/core/_methods.py:49...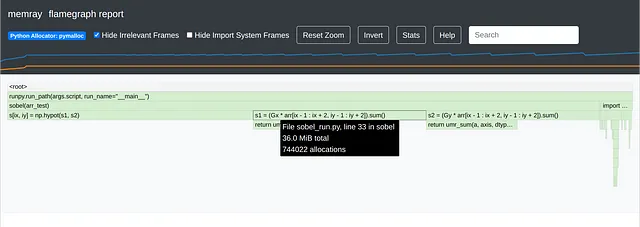
Beta版本和一个错误
现在我们有了一个可行的Alpha版本和一些分析函数,我们将利用SciPy库获得一个更快的版本。
from typing import Tupleimport numpy as npfrom numpy.core.multiarray import normalize_axis_indexfrom numpy.typing import NDArrayfrom scipy.signal import convolve2ddef sobel_conv2d( arr: NDArray, axes: Tuple[int, int] = (-2, -1)) -> NDArray: if arr.ndim != 2: raise NotImplementedError if any( normalize_axis_index(ax, arr.ndim) != i for i, ax in zip(range(2), axes) ): raise NotImplementedError # 将卷积核创建为一个复数数组。这样我们可以使用np.abs而不是np.hypot来计算幅值。 G = np.array( [[-1, 0, 1], [-2, 0, 2], [-1, 0, 1]], dtype=arr.dtype, ) G = G + 1j * np.array( [[-1, -2, -1], [0, 0, 0], [1, 2, 1]], dtype=arr.dtype, ) s = convolve2d(arr, G, mode="same") np.absolute(s, out=s) # 原地绝对值 return s.real
sobel_timed = timeit(sobel_conv2d)sobel_timed(arr_test)# 14.3 ms ± 1.71 ms per loop (mean ± std. dev. of 10 runs)好多了!但是这样对吗?
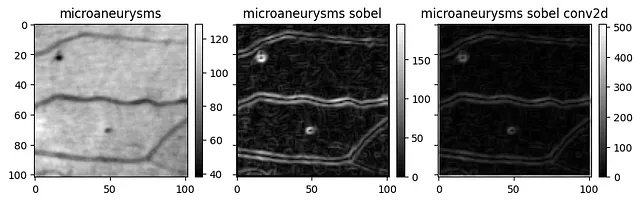
这些图片看起来非常相似,但是如果你注意颜色刻度,它们是不同的。运行测试会标志出一个较小的平均误差。幸运的是,我们现在已经能够很好地检测出定量和定性的差异。
在调查这个错误之后,我们将其归因于不同的边界条件。查看 convolve2d 文档告诉我们在应用卷积核之前,输入数组会被填充零。在alpha版本中,我们填充了输出!
哪个是正确的?可以说SciPy的实现更有道理。在这种情况下,我们应该采用新版本作为标准,如果需要的话,修复alpha版本以使其与之匹配。这在科学软件开发中很常见:如何更好地完成任务的新信息会改变标准和测试。
在这种情况下,修复很简单,用零填充数组然后再进行处理。
def sobel_v2(arr: NDArray, axes: Tuple[int, int] = (-2, -1)) -> NDArray: # ... arr = np.pad(arr, (1,)) # 填充后的形状为 (nx + 2, ny + 2) s = np.zeros_like(arr) for ix in range(1, arr.shape[0] - 1): for iy in range(1, arr.shape[1] - 1): s1 = (Gx * arr[ix - 1 : ix + 2, iy - 1 : iy + 2]).sum() s2 = (Gy * arr[ix - 1 : ix + 2, iy - 1 : iy + 2]).sum() s[ix - 1, iy - 1] = np.hypot(s1, s2) # 调整索引 return s一旦我们纠正了函数,我们可以更新依赖它们的标准和测试。
最后的想法
我们看到了如何将本文中探讨的一些软件开发思想付诸实践。我们还看到了一些可以用来开发高质量、高性能代码的工具。
我建议你在自己的项目中尝试一些这些想法。特别是,练习对代码进行性能分析和改进。我们编写的Sobel滤波函数非常低效,我建议试着改进它。
例如,尝试使用像Numba这样的即时编译器对CPU进行并行化,将内部循环移植到Cython中,或者使用Numba或CuPy实现一个CUDA GPU函数。欢迎查看我关于使用Numba编写CUDA内核的系列文章。






